
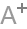
虽然很多人都知道用母乳喂养小宝宝好处多,但是由于各方面的原因,很多妈妈却忽视了这件事的重要性。至于这件事情到底有多重要,我们一起来看看今年2月份发表在顶级期刊上的三篇文章就知道了。
这三篇文章,有两篇发表在《科学》上,另一篇发表在《细胞》上。它们阐述了儿童营养不良与肠道微生物之间的关系,研究人员把营养不良儿童的肠道菌群移入小鼠体内,使小鼠也发育不良,当研究人员给小鼠补充母乳中的糖类或是某种微生物之后,小鼠便恢复正常生长发育。
这三个研究告诉各位妈妈:宝宝吃的可不仅仅是奶,还有很重要的微生物!所以能用母乳喂养的话呢,就尽量不要用奶粉啦。要知道,在全世界范围内的五岁以下婴幼儿死亡案例中,将近一半都是由营养不良引起的,而恢复充足的营养摄入量也只能起到一定的作用。
对人类来说,从怀孕到出生后三岁之间对于成长发育是至关重要的。数十年来对人类健康和疾病的研究都表明,在这个阶段中,环境影响导致的疾病可能会伴随之后的生活。例如,早期阶段的营养不足或是营养过剩都可能导致终身的新陈代谢和心血管问题。由于肠道菌群在肠道代谢方面的共生机制,它的成分的改变常被认为是导致疾病的“源头”。
我们吃的食物会影响肠道微生物的生活环境,而每种微生物也都有优先支持它生长的最佳代谢环境。举例子来说,富含植物纤维的饮食结构塑造的肠道菌群和富含动物脂肪的饮食习惯相比有着巨大的差异。反过来呢,菌群也会帮助消化其他难消化的成分,比如纤维,分解后可以作为肠细胞生长的能量提供者,并且可以塑造健康的免疫系统。
那么当儿童营养不良时,肠道菌群会发生什么呢?在第一篇文章中,Blanton和其他研究人员解决了这个问题。他们收集了发育正常的和不同程度营养不良的马拉维地区儿童的粪便样本,提取出其中的菌群并做了分析。首先使用正常发育儿童的菌群状况做一个对比模型,然后把整体样本菌群放入模型中,于是研究人员发现,体重增长不正常的儿童肠道内有不成熟的菌群存在。
Blanton等人接下来研究了肠道菌群对儿童发育影响的不同之处。他们将细菌从粪便样本中分离出来,移植到无菌小鼠的肠道内,然后用缺乏营养的食物饲养小鼠,模拟典型的马拉维地区的饮食结构。
在几周后,小鼠持续按照这个模式生长(我们将其称为营养不良型小鼠),与携带正常菌群的对照组小鼠相比,它们的体重增长明显受限。但是,如果把两组小鼠放在同一个笼子里,对照组小鼠携带的细菌会迅速转移到营养不良的小鼠体内,使它们的体重恢复到健康水平。研究人员对菌群中的两种细菌做了鉴定,分别是活泼瘤胃球菌和共生梭菌,它们对营养不良小鼠体重的增长起到了作用。
两组小鼠的发育情况
第二篇文章中,Schwarzer等人研究出了肠道菌群如何在儿童发育早期系统地辅助生长发育。他们比较了正常发育的小鼠和营养不良小鼠,发现菌群可以通过促进胰岛素生长因子蛋白的合成来帮助身体发育。[2]如果小鼠处于营养缺乏的饮食结构中,细菌对这个生长因子的刺激能够一定程度上改善不良的生长状况。但不是所有细菌都能以这种方式促进生长发育,比如说植物乳杆菌。这些实验结果告诉我们,每种微生物对它的宿主都有着不同的影响,但是有一点需要注意的是,对小鼠生长发育有益的微生物不一定对人类也有同样的作用。
Charbonneau和他的同事们发现,有些生长发育是依赖于微生物刺激的,但是培育特殊菌株并不是完成这种刺激的唯一方式[3]。他们对营养不良儿童母亲的母乳做了研究,发现她们母乳中乳类低聚糖的量明显较少。这种糖类非常有趣,它们只存在于人乳中,在牛奶中是没有的。
为了搞清楚这种糖类是否能够促进微生物依赖型生长,研究人员使用和Blanton等人相似的方法让小鼠产生营养不良的症状,在其饮食中增加一种特殊的唾液酸乳类低聚糖,可以观察到明显的体重增加现象。反之,如果使用其他糖类或者是将其添加到正常生长发育小鼠的肠道内,则没有任何效果。
虽然加入糖类不会明显地改变任何一种微生物的数量,但是它会增加淀粉代谢和其他代谢过程中微生物基因的转录。研究人员总结出,它的增加可能是整体群落中一系列复杂反应导致的结果,必须开展额外的研究才能搞清楚这些反应的过程和机理。
人工添加可以帮助一些不能从母乳中获取乳类低聚糖的儿童更好地发育。但是,人工合成这种低聚糖是有困难的,所以目前这种方法不能够广泛应用。
多个研究成果都证明饮食结构、肠道微生物和健康三者之间有着千丝万缕的联系。当我们调整饮食结构时,可能会影响体内数万亿细菌的生长。
文中提到的三篇文章使我们在肠道菌群如何导致营养不良和生长发育缺陷方面有了更清晰的认识,但是他们并没有找到能够解决这个问题的灵丹妙药。此外,因为营养不良在不同的群体间有不同的原因,所以我们应该开展更多的基于人类群体的研究,探讨如何优化饮食结构,重视菌群的有益影响。
参考文献:
[1] Blanton, L. V. et al. Science 351, aad3311 (2016).
[2] Schwarzer, M. et al. Science 351, 854–857 (2016).
[3] Charbonneau, M. R. et al. Cell 164, 859–871 (2016).